Microbial community succession characteristics in an anaerobic baffled reactor during phenol gradient acclimation
-
摘要:
厌氧消化是工业废水和城市废水处理过程中常用的技术之一. 由于废水中的苯酚会影响厌氧消化系统的稳定性,因此在使用厌氧消化工艺前需要使用苯酚对活性污泥进行驯化,从而降低其抑制作用. 本文考察了厌氧折流板反应器在进行苯酚梯度驯化时各隔室出水的化学需氧量(COD)、pH、苯酚及沼气中氢气分压的变化情况. 运用Illumina高通量测序平台分析了细菌和古菌群落的演替过程,并使用气相色谱与质谱联用仪(GC–MS)和高效液相色谱仪(HPLC)对苯酚厌氧降解产物分别进行了定性和定量分析. 结果表明,使用葡萄糖作为唯一进水碳源时第1隔室出水的pH在5.0~6.8之间波动. 当进水中加入苯酚后该隔室出水pH有所上升. 约90%的COD和苯酚在前两个隔室内降解,但后续隔室的COD始终保持在100 mg·L−1左右,因此推测最终出水中含有未降解物质. 使用葡萄糖和苯酚作为进水碳源时细菌和古菌群落的α生物多样性指数高于使用单一进水碳源. 苯酚的加入导致细菌群落的优势菌科Streptococcaceae和Enterobacteriaceae被Syntrophaceae取代;古菌群落的优势菌科Methanobacteriaceae 则被Woesearchaeales取代. 冗余分析显示,细菌Syntrophaceae科和古菌Woesearchaeales科分别与苯酚和乙酸的相关性较高. 产物定性分析确认了最终出水中的难降解物质为丙酸. 热力学计算表明反应器内过高的氢气分压是导致丙酸难以降解的原因.
Abstract:Anaerobic digestion (AD) is extensively used for treating both industrial and municipal wastewater owing to its cost-efficiency and eco-friendly nature. However, phenol in polluted effluents can destabilize AD performance. To increase phenol tolerance to AD microbial communities, stepwise acclimatization is commonly employed. This method also aids in selecting microorganisms well-suited for phenol degradation. In this study, anaerobic sludge acclimatization was conducted using a four-chamber anaerobic baffled reactor (ABR). Initially, the reactor was fed with glucose as the sole carbon source. Phenol levels in the influent increased stepwise from 100 to 300 mg·L−1. In the final experimental phase, the reactor processed wastewater containing 780 mg·L−1 of phenol as the sole substrate, without glucose. We analyzed hydrogen partial pressure, pH, chemical oxygen demand (COD), and phenol concentration to examine changes in reactor performance. The Microbial community dynamics were investigated using Illumina high-throughput sequencing technology. Phenol degradation intermediates were performed using gas chromatography/mass spectrometry (GC–MS), with their quantities measured by high-performance liquid chromatography. The results showed a decrease in hydrogen partial pressure from 5 to 20 Pa in the ABR from Chamber 1 to Chamber 4. The pH levels in the first chamber fluctuated from 5.0 to 6.8 with glucose alone and increased with phenol addition. Nearly 90% of COD and phenol were degraded in the initial two chambers. However, COD levels remained at 100 mg·L−1 in the last two chambers, suggesting the presence of nondegradable components in the effluent. α-diversity analysis indicated Shannon, Simpson, and Chao1 indices for bacterial and archaeal communities with glucose and phenol as carbon sources in the feed. Acclimatization significantly altered the microbial community structure, shifting dominant families from Streptococcaceae and Enterobacteriaceae to Syntrophaceae, and archaeal families from Methanobacteriaceae to Woesearchaeales. Redundancy analysis (RDA)linked Syntrophaceae and Woesearchaeales to phenol degradation and acetotrophic processes, respectively. GC–MS analysis revealed propionic acid as the only effluent component. The randomized methyl–malonyl–CoA pathway and the C-6-dismutation pathway are two known routes for propionate degradation. However, the ΔG' values for these reactions were consistently greater than −20 kJ·mol−1, indicating that propionate degradation did not occur under the experimental conditions. From a thermodynamic point of view, high hydrogen partial pressure levels in the reactor affected the propionate degradation process and also inhibited sludge bioactivity. Syntrophobacter and Smithella are responsible for degrading propionate. The relative abundance of these two families slowly increased from 0.35% to 6.9% in the ABR from Chamber 1 to Chamber 4. These findings account for the inadequate efficiency of COD removal observed in the effluent.
-
苯酚是焦化、冶金、炼油、石油化工等工业废水中的主要有机污染物,特别在煤炭加工和炼焦废水中其占比可达60%[1−3]. 苯酚具有中等强度的化学毒性,能够引起生物体蛋白质变性和基因突变,超标排放苯酚废水会对生态系统和人类健康造成严重的威胁[4].
生物法是一种较为普遍的、经济有效的处理含酚废水的技术方法. 其中厌氧生物处理法具有抗毒性冲击能力强、有机负荷高、耗能少、运行成本低等优点,因此常被设计为含酚废水处理过程中的第一道工艺[5−7]. 目前已知的参与常温厌氧条件下苯酚降解反应的细菌多分布在脱硫杆菌门(Desulfobacterota). 该菌门中的互营菌科(Syntrophaceae)、互营杆菌科(Syntrophobacteracea)和Smithellaceae在与耗氢产甲烷菌共生时可以互营代谢苯酚并产生短链脂肪酸和氢气[8]. 在这一过程中,苯酚首先通过羧化和脱羟基反应被转化为苯甲酸,随后苯甲酸被还原为环己烷羧酸或环己烯羧酸并通过β氧化过程被最终分解为乙酸、氢气和二氧化碳[9]. 由于高浓度的乙酸和氢气会抑制降解过程,因此只有当苯酚降解菌与产甲烷菌形成稳定的共生群落后才能实现苯酚的持续降解[10]. 然而高苯酚负荷往往会破坏稳定的微生物群落,导致降解效率下降. 为了保证厌氧消化工艺高效、长久的运行,在使用厌氧工艺处理毒性废水之前往往需要对反应器内的活性污泥进行驯化[11−13]. 厌氧折流板反应器 (Anaerobic baffled reactor, ABR) 通过内置的竖向折流板将反应器分隔为若干个串联的隔室. 这种结构使得毒性物质往往只对前端隔室的污泥微生物群落产生冲击,而后端隔室受到的干扰较小[14],因此该反应器常用于活性污泥驯化研究[15−16].
分子生物学技术是研究活性污泥驯化过程中微生物群落演替规律的重要手段之一. 然而早期技术存在的测序通量低、精度差以及可检测微生物种类少的缺点在一定程度上限制了相关研究的开展[17−18]. 第二代测序(Next generation sequencing, NGS)技术的快速普及使得研究者能够以低廉的成本快速获得高质量的基因组数据,相关研究也因此呈现出指数级增长[19−20]. 近些年,计算机硬件性能的提升以及生物信息分析软件功能的不断完善进一步推动了微生物群落结构、功能与降解过程相关性研究向更深层次发展.
本研究采用Illumina高通量测序平台,对苯酚驯化条件下ABR中微生物群落的演替过程进行了分析,着重考察了环境因子变化对优势菌群的影响,并对苯酚厌氧降解产物及其降解热力学条件进行了分析,本研究结果为特异性污染物降解菌群的筛选及提升污染物降解效率提供理论依据.
1. 材料与方法
1.1 反应器参数与进水条件
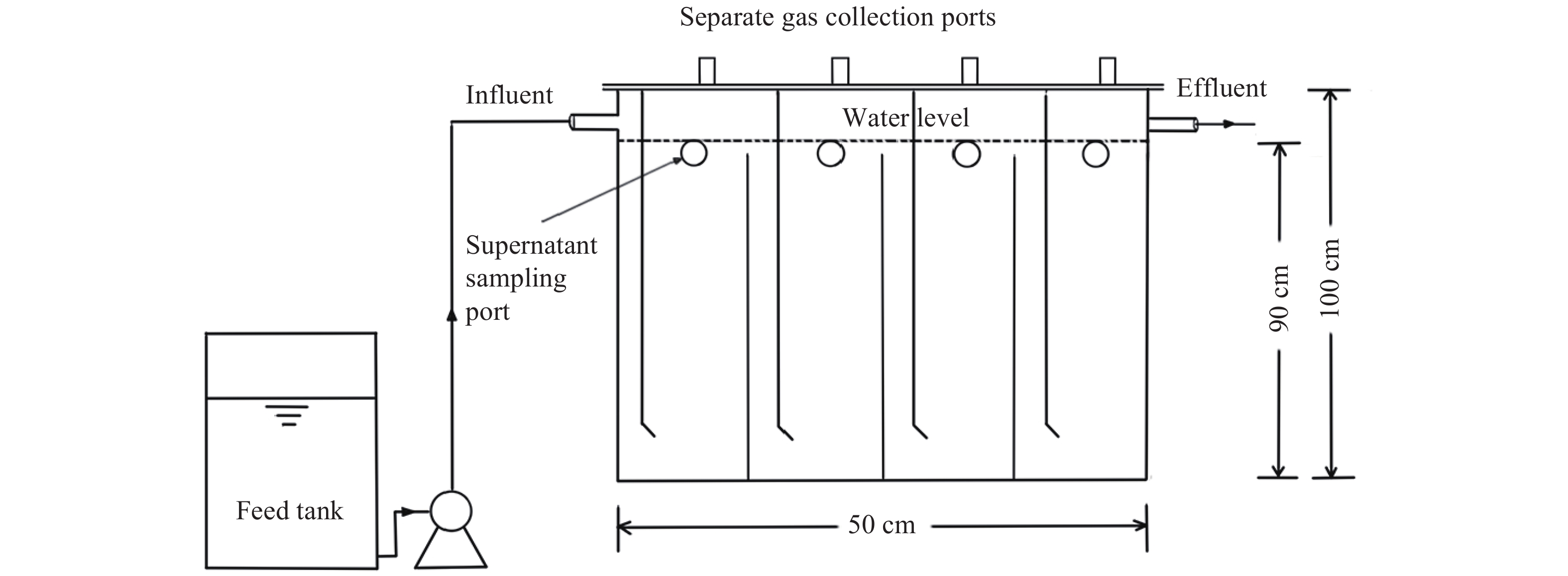
实验用ABR包括4个大小相同的隔室,每个隔室体积为16.9 L,反应器总体积67.5 L (图1). 反应器整体用保温材料包裹,使用蠕动泵控制进水流量. 反应器水力停留时间为45 h,运行温度28 ± 2 ℃,进水COD的质量浓度为2000 mg·L−1.
接种污泥取自北京市高碑店污水处理厂厌氧发酵罐和好氧池的回流污泥. 模拟废水使用葡萄糖和苯酚作为碳源, COD、氮元素(氯化铵)和磷元素(磷酸二氢钾)质量比为500∶5∶1,同时添加0.66 g·L−1碳酸氢钠和微量元素培养液[21].
1.2 仪器与试剂
NOVA 60(美国MERCK公司)多参数水质分析仪;PHS-25酸度计(中国雷磁公司);GCMS-QP2010-plus气相色谱与质谱联用仪(日本岛津公司);Prominence高效液相色谱仪(日本岛津公司);Milli-Q A 10超纯水处理系统(美国密理博公司);Gasboard-3100P便携式气体分析仪(中国锐意自控系统有限公司).
甲醇、乙腈、磷酸二氢钾、磷酸均为色谱纯(美国赛默飞公司). 脂肪酸混合标准溶液、苯甲酸和苯酚标准物质购于美国AccuStandard公司. Strata-X型固相萃取柱来自美国Phenomenex公司. 其他试剂等级均为分析纯(中国国药集团化学试剂有限公司).
1.3 水样处理与检测方法
1.3.1 水样处理方法
使用混合纤维素微孔滤膜(孔径0.45 μm)过滤水样约40 mL,留取30 mL用于pH、COD、苯酚和高效液相色谱(HPLC)检测.
采用固相萃取法纯化水样,具体操作为:取10 mL过滤后水样,加盐酸调节pH至2.0后备用. 依次使用甲醇和超纯水淋洗萃取柱,随后将2 mL酸化水样加入到萃取柱,待水样全部流出后加1 mL水清洗,然后真空干燥1 min,取出后使用1 mL甲醇淋洗,收集洗脱液供气相色谱进样.
1.3.2 COD和苯酚检测方法
采用重铬酸钾法测定COD,采用4-氨基安替比林比色法测定苯酚浓度. COD操作过程为:取过滤水样10 mL加入到预装试剂比色管中并消煮2 h,待比色管冷却后使用水质分析仪检测;苯酚检测流程按照试剂盒操作手册进行.
1.3.3 GC–MS定性分析方法
气相色谱柱为ZB-FFAP(30 m × 0.32 mm ×0.25 μm,Phenomenex,美国). 色谱条件:初始温度80 ℃保持1 min,随后以每分钟8 ℃的速度程序升温至220 ℃,保持3 min;自动进样器设置进样量为1 μL,进样方式为不分流进样,进样口温度为180 ℃;载气为高纯氦气,气体柱流量为1.5 mL·min−1. 质谱条件:电子轰击离子源(EI),电子能量为70 eV,SCAN模式扫描范围45~300 Da,离子源和接口温度均为240 ℃,谱图数据库为NIST14.
首先使用上述测试条件进样甲醇2~3次,待基线平稳后再进行样品检测. 降解产物定性采用基于相似度匹配的质谱图库搜索并结合标准品验证保留时间的方法.
1.3.4 HPLC定量分析方法
液相色谱柱为Acclaim Organic Acid (250 mm × 4.0 mm,Thermo Fisher Scientific, 美国). 色谱梯度洗脱条件为: 流动相A 为20 mmol·L−1磷酸二氢钾溶液 (使用磷酸调节pH 至2.5),流动相B为乙腈. 梯度洗脱程序:0~3 min, 100% A; 3~25 min 80% A. 柱温35 ℃, 流速1 mL·min−1,进样量 10 µL,检测波长为210 nm. 分别配制5个浓度的混合脂肪酸、苯甲酸和苯酚标准溶液,采用外标法定量.
1.4 试验设计与污泥样品采集
在驯化实验开始前使用葡萄糖作为碳源配制模拟废水,维持反应器运行6个月. 当出水COD基本稳定后开始正式试验.
苯酚驯化试验共分为4个阶段,每个阶段进水中的葡萄糖和苯酚比例均不相同,但进水COD始终保持在2000 mg·L−1左右(表1). 反应器共计运行158 d,每2天测定所有隔室水样的COD、pH和苯酚含量. 从第159天开始,连续5天对所有隔室水样进行定性和定量分析.
表 1 不同阶段ABR运行条件Table 1. Operating conditions of ABR across different phasesStage Glucose
concentration/(g·L−1)COD equivalent of
glucose/(g·L−1)Phenol
concentration/(g·L−1)COD equivalent of
phenol/(g·L−1)Time/d I 2.00 2.02 0 0 62 II 1.75 1.75 0.10 0.26 26 III 1.24 1.24 0.30 0.77 46 IV 0 0 0.78 1.99 22 污泥取样使用玻璃管在反应器的底部、中部和上部各取10 mL左右的泥水混合物,混匀过滤后将滤纸与污泥一同密封至离心管并放置在−80 ℃冰箱保存. 每个阶段分别在4个隔室中取污泥样品1份,试验共取样16份. 操作过程中所用滤纸及器皿均经过高温灭菌,污泥混匀、过滤及密封操作在超净工作台中进行.
第I阶段污泥样品命名为对照组(Control group, CG),每个隔室样品名依次为CG.1,CG.2,CG.3和CG.4;第II阶段进水中苯酚(Phenol, Phe)质量浓度为100 mg·L−1,样品名依次为Phe100.1~Phe100.4;第III、IV阶段样品名依此规则,分别为Phe300.1~Phe300.4和Phe780.1~Phe780.4.
1.5 DNA提取与高通量测序
污泥DNA提取及第二代测序工作委托北京诺禾致源科技股份有限公司完成. 污泥样品首先经液氮冷冻后研磨成粉末,采用FastDNA SPIN Kit for soil (MP Biomedicals, 美国)试剂盒按照操作手册提取总DNA;采用1%琼脂糖凝胶电泳定性检测DNA,采用分光光度计对DNA浓度和纯度进行检测.
使用Illumina HiSeq PE250平台对聚合酶链式反应(PCR)的扩增产物进行高通量测序. 细菌和古菌分别设计引物对16s rRNA基因的V4可变区进行扩增. 细菌引物为515F(5′-GTGCCAGCMGCCGCGGTAA-3′)和806R(5′-GTGCCAGCMGCCGC-GGTAA-3′)[22];古菌引物为Arch519F(5′-CAGYMGCCRCGGKAAHACC-3′)和Arch806R (5′-GGACTACNSGGGTMTCTAAT-3′)[23].
1.6 数据分析
测序原始数据使用QIIME 2-
2022.2 进行分析[24]. 具体分析流程为:使用q2-demux插件去除原始数据中的标签和引物序列;去除低质量序列,所有样品序列最终长度为180 bp;使用DADA2[25]进行序列去噪和拼接并生成扩增序列变体(Amplicon sequence variants,ASV);多样性计算采用q2-diversity,细菌和古菌分别使用27318 和11751 条序列数进行抽平,并绘制样本稀释曲线,观察曲线基本平稳,说明取样充足且合理. 物种注释采用q2-feature-classifier插件,使用朴素贝叶斯算法分类器(Naive Bayes classifier),参考数据库为Silva 16s rRNA全长数据库[26](Silva 138 99% OTUs full-length sequences).数据统计使用R语言vegan数据包完成[27]. 使用基于转化的冗余分析法(Transformation-based redundancy analysis,tb-RDA)展示物种丰度与环境因子之间的关系. 3个环境因子分别为苯酚浓度,pH和COD',其中COD' 表示在总COD中扣除苯酚的贡献,所有环境因子均为7 d数据的平均值,苯酚浓度和COD' 采用对数转换,菌种丰度数据使用Hellinger法转换. 使用vegan包中的anova命令对tb-RDA结果进行置换检验,使用anova.cca命令进行环境因子显著性检验,置换次数均为999.
2. 结果与讨论
2.1 水质变化分析
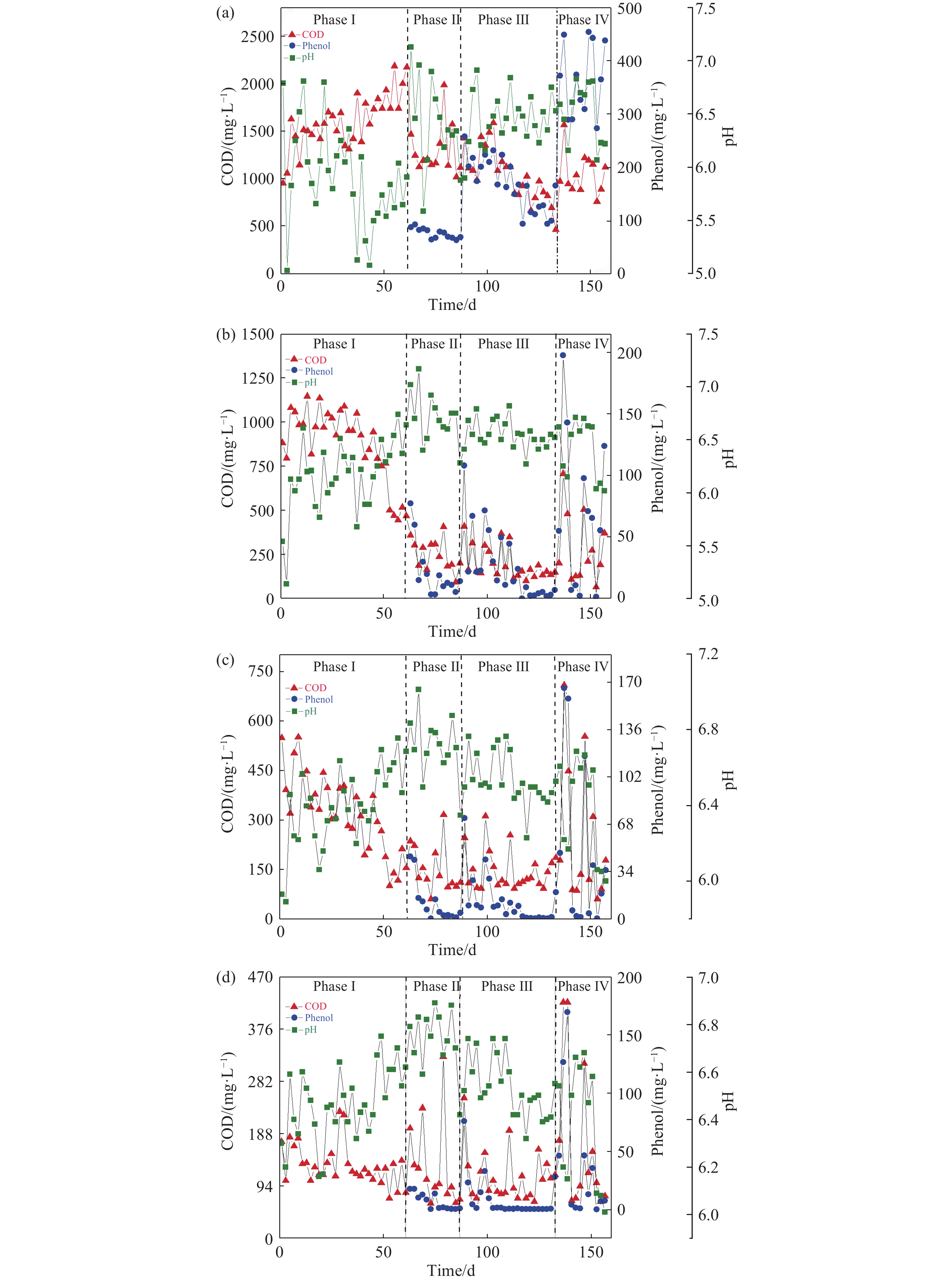
在驯化试验的第I阶段,第1隔室水质变化结果如图2(a)所示. 此时隔室内pH波动较大,最低值仅为5.0左右,说明酸化反应进行的较为剧烈. 然而由于过低的pH会抑制产甲烷菌生长,因此该阶段的COD去除率仅为20%左右. 在后续隔室中pH稳定在6.0~6.5之间,说明有机酸的不断降解,COD也呈现稳步下降的趋势(如图2(b), 2(c), 2(d)所示).
在第II、III阶段,随着进水中的葡萄糖逐步被苯酚替代,所有隔室内的pH均有所升高. 推测这是由于苯酚的生物可降解性弱于葡萄糖,因此酸性物质产生的较慢,从而导致pH波动减小. 而此时的pH更有利于厌氧反应的进行,第1、2隔室的COD去除率已经达到85%以上,苯酚基本完全降解,进入到第3、4隔室中的COD基本保持在100 mg·L−1的水平.
第IV阶段以苯酚作为唯一进水碳源,此时第1、2隔室降解了90%以上的苯酚并去除了95%以上的COD,在第3、4隔室中苯酚和COD基本与上一阶段相同,但是pH却较前三个阶段出现了下降,推断在出水中存在难降解的酸性物质. 此外在污泥取样时发现,由于第3、4隔室的COD长期处于较低水平,其污泥生物量远低于前端隔室.
2.2 微生物群α多样性分析
细菌和古菌群落的Shannon、Simpson和Chao1指数如表2所示. 其中Shannon和Simpson指数同时考量物种丰富度和均匀度对群落α多样性的影响,但后者更易受到均匀度的影响;Chao1指数对稀有物种十分敏感,但与物种丰富度和均匀度无关. 在第I阶段,水力冲击负荷最大的第1隔室中细菌和古菌群落的α多样性均为最低,随着后续隔室中水力负荷的逐步下降以及可利用碳源的增多,菌群的α多样性逐步升高,此时第4隔室中菌群的Shannon和Simpson指数虽略低于第3隔室,但是Chao1指数却最大. 在第II阶段,苯酚的加入使得进水碳源更加丰富,此时第1隔室中细菌和古菌群落的α多样性指数均有所升高,但第4隔室的α多样性却明显下降. 在第III阶段所有隔室菌群的α多样性均较上一阶段有所增加,第IV阶段以苯酚作为唯一进水碳源,此时的α多样性指数重新恢复到与第一阶段接近的水平. 因此使用多种有机碳源混合培养可以显著的提升菌群的α多样性,从而提升ABR系统的稳定性.
表 2 不同污泥样品的α多样性分析Table 2. α-diversity analysis of different sludge samplesSamples Bacterial community Archaeal community Shannon Simpson Chao1 Shannon Simpson Chao1 CG.1 2.01 0.65 284 3.42 0.90 404 CG.2 3.37 0.89 230 3.96 0.92 415 CG.3 5.37 0.99 673 4.48 0.97 394 CG.4 5.13 0.98 716 4.25 0.95 632 Phe100.1 2.87 0.80 305 3.59 0.90 510 Phe100.2 3.36 0.89 317 4.29 0.95 564 Phe100.3 4.09 0.95 416 4.65 0.97 589 Phe100.4 3.92 0.92 383 4.82 0.98 486 Phe300.1 5.44 0.98 1027 3.68 0.87 1100 Phe300.2 5.70 0.97 1111 4.43 0.88 1620 Phe300.3 5.68 0.98 860 4.91 0.94 1489 Phe300.4 4.14 0.94 292 5.78 0.99 1585 Phe780.1 3.57 0.90 402 2.96 0.84 370 Phe780.2 4.46 0.95 608 4.37 0.97 458 Phe780.3 5.22 0.98 865 4.17 0.96 585 Phe780.4 5.28 0.99 807 4.45 0.97 580 2.3 微生物群落演替规律分析
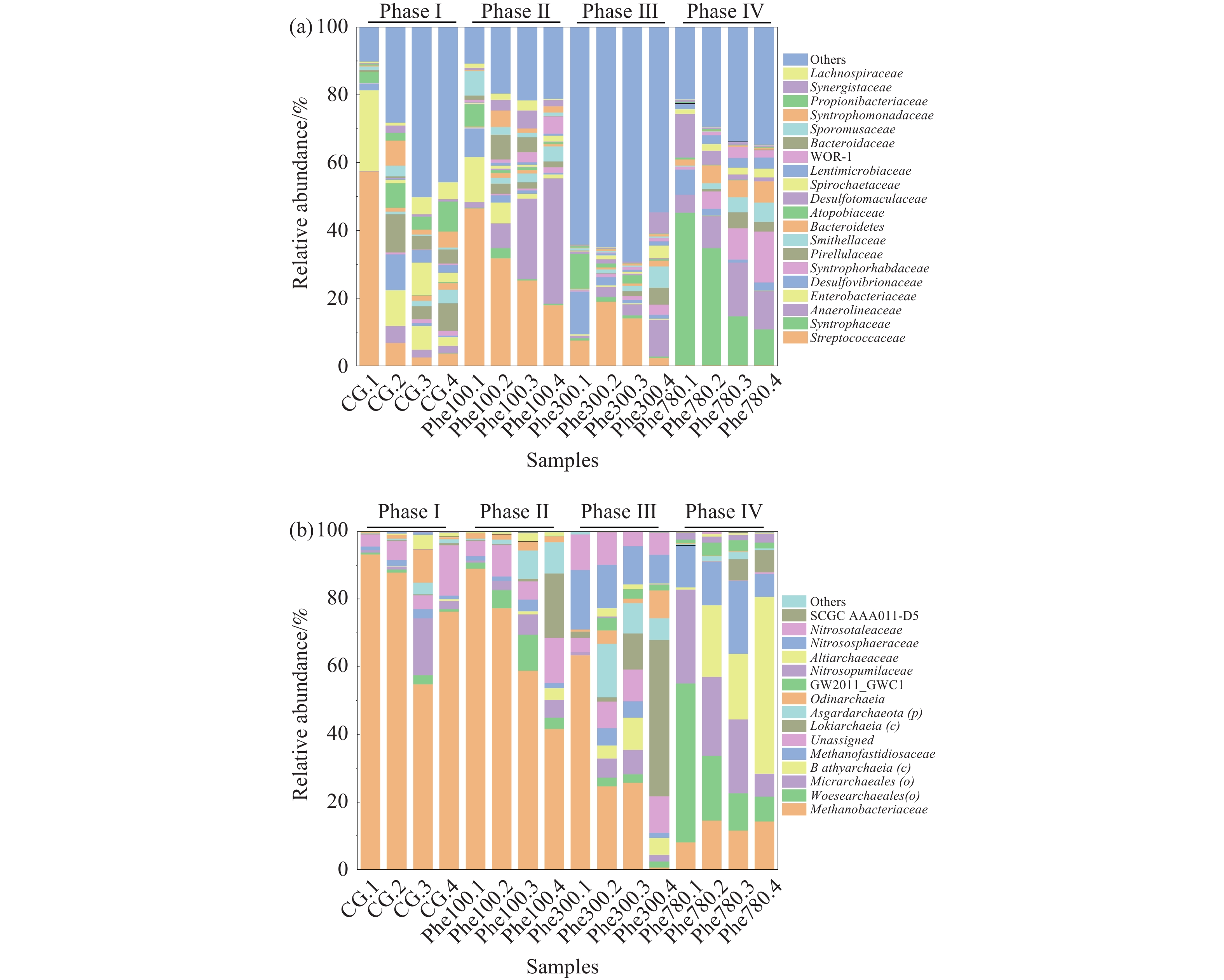
细菌群落测序共获得
584656 条序列,分别归属于46个门,461个科. 选取相对丰度占比最高的20个科,将其他科合并为Others,如图3(a)所示. 在第I阶段,链球菌科(Streptococcaceae)和肠杆菌科(Enterobacteriaceae)是第1隔室中的主要菌群(相对丰度分别为57.2%和23.9%),该科菌种主要以葡萄糖为生长碳源 [28]. 在后续隔室中以上两种菌科的丰度迅速下降,具有硫酸盐还原能力的脱硫弧菌科(Desulfovibrionaceae)和pirellulaceae相对多度明显升高. 在第II阶段,链球菌科和肠杆菌科的丰度持续下降,而厌氧绳菌科(Anaerolineaceae)的丰度有所上升. 在第III阶段,仅链球菌科仍然具有较高的丰度,而丰度较低的菌种占比已达到54.7%~69.3% (Others),这与该阶段细菌群落生物多样性较高的结论一致. 在第IV阶段,具有苯酚降解能力的互营菌科(Syntrophaceae)成为各隔室中的主要菌种,在第1隔室中其丰度已经达到45%,此外与丙酸降解相关的互营杆菌科(Syntrophobacteracea)和Smithellaceae科的丰度也有所上升,因此推断降解过程有丙酸产生[29].古菌群落测序共获得
35350 条序列,分别归属9个门. 由于目前古菌数据库的分类信息很难将其注释到科水平,因此对于未注释到科水平的古菌使用该物种目或纲水平名称代替. 其中Woesearchaeales和Micrarchaeales为菌目名称,Bathyarchaeia和Lokiarchaeia为菌纲名称,Asgardarchaeota为菌门名称,其他古菌使用科名称表示. 将相对丰度小于0.1%的数据合并为其他(Others)之后共得到14个科,如图3(b)所示. 在第I阶段,甲烷杆菌科(Methanobacteriaceae)是所有隔室中丰度最高的菌种(丰度最大值为93.1%),由于该科属于氢营养型产甲烷菌,因此可以推测该阶段氢气产量较大,此外Unassigned(未分类菌种)丰度最大值接近15%(第4隔室)说明在厌氧微生物群落中还有存在较多的未分类古菌. 在第II阶段,甲烷杆菌科的丰度开始下降,之前丰度仅为1%左右的Asgardarchaeota在第3、4隔室中升高至8%以上,Woesearchaeales和Micrarchaeales的丰度也出现了一定的增长. 在第III阶段,甲烷杆菌丰度由第1隔室的63.3%快速下降至第4隔室的0.6%,Nitrososphaeraceae和Nitrosotaleaceae在此阶段丰度由先前的0.1%快速增长到10%左右. 在第IV阶段,Methanobacteriaceae的优势地位已经被Woesearchaeales和Micrarchaeales属取代,两者在第1隔室中丰度之和达到74.8%;在后续隔室中Bathyarchaeia的丰度快速增加,在第4隔室中其丰度达到52.3%,此外Methanofastidiosaceae的丰度也出现了2~4倍的提升,而未分类菌种的丰度迅速下降至0.5%以下. 由于本研究仅检测到极少量的乙酸营养型产甲烷菌Methanotrichaceae(原分类名为甲烷鬃毛菌科Methanosaeta,丰度约为0.1%),因此推测优势菌属Woesearchaeales和Micrarchaeales参与了乙酸降解过程.2.4 微生物群落与环境因子的关系
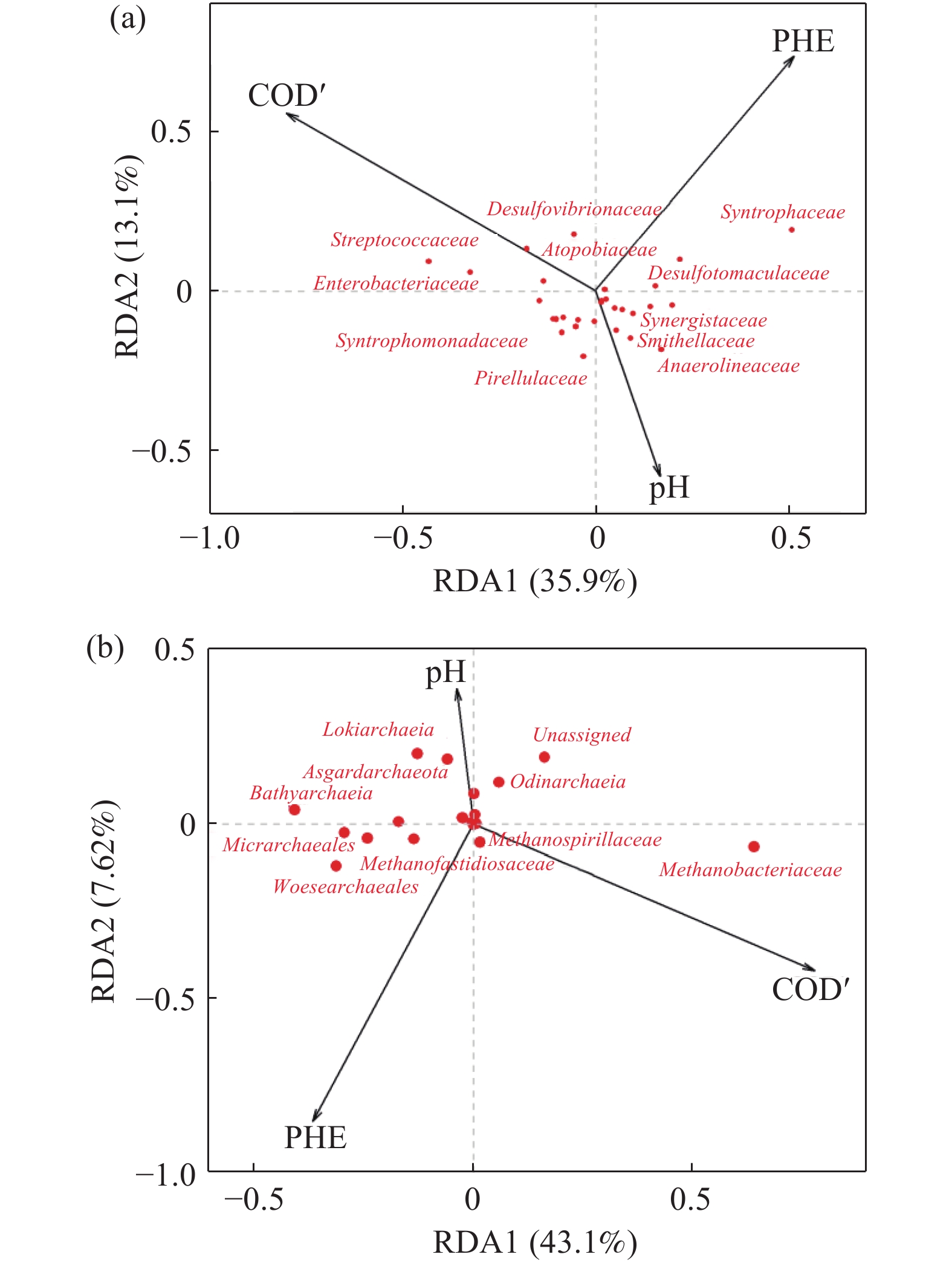
在方差解释率方面,3个环境变量分别解释了细菌群落和古菌群落51.8%和52.3%的总体方差(校正后数值分别位39.7%和40.4%);RDA模型的显著性达到非常显著水平(p = 0.001);在单一环境变量显著性检验方面,COD’对细菌群落的影响达到非常显著水平(p = 0.003),对古菌群落影响达到极其显著水平(p = 0.001);苯酚对细菌和古菌群落的影响均达到显著水平(p 值分别为0.022,0.030);由于本研究使用碳酸氢钠对进水pH进行了调节,因此检验结果显示pH对细菌的影响仅达到一般显著水平(p = 0.083),对古菌群落则没有显著影响(p = 0.617).
细菌和古菌群落在科水平的RDA分析结果如图4所示(分别显示20个细菌科和14个古菌科),图中的横纵轴RDA1与RDA2分别代表典范轴中对响应变量(物种数据)的方差解释率最高的两个轴,括号中注释的数值表示解释变量(环境因子)对响应变量的解释率. 在细菌群落中,链球菌科(Streptococcaceae)、肠杆菌科(Enterobacteriaceae)和厌氧绳菌科(Anaerolineaceae)趋向分布在高COD’的环境中,这与该菌科具有将葡萄糖转化为乙酸的功能相一致,而苯酚对其分布未产生影响;互营菌科(Syntrophaceae)和Desulfotomaculaceae具有将苯酚降解为乙酸的功能[29−30],因此趋向于分布在高苯酚的环境中,但与COD’相关性较弱;Desulfovibrionaceae具有将多种有机物降解为乙酸的功能[31],由于其分布与苯酚和COD’均呈正相关,因此推断该菌种可以同时利用来自于葡萄糖和苯酚分解产生的有机酸;Anaerolineaceae、Pirellulacea、Smithellaceae和Syntrophomonadaceae分布在低苯酚、低COD’和高pH环境,这些菌种在耗氢产甲烷菌存在的情况下可以分解有机酸[32−34],但仅适合在底物浓度较低的环境下分布. 聚集分布在坐标系中部且距离中心点较近的菌群可能在整个环境中均匀分布或只分布在环境梯度的中值区域.
![]() 图 4 微生物群落结构与环境因子间的RDA分析(红点表示群落中的优势菌科,黑色箭头表示环境因子,箭头的长度表示环境因子的影响力,箭头方向与红点的夹角表示微生物群落结构与该环境因子的相关性). (a)细菌群落; (b)古菌群落Figure 4. RDA of microbial community structure and environmental factors (red circles: dominant families; black arrows: the environmental factors; arrow length: the magnitude of the environmental factors; arrow direction: the correlation between the environmental factors and the microbes): (a) bacterial community; (b) archaeal community
图 4 微生物群落结构与环境因子间的RDA分析(红点表示群落中的优势菌科,黑色箭头表示环境因子,箭头的长度表示环境因子的影响力,箭头方向与红点的夹角表示微生物群落结构与该环境因子的相关性). (a)细菌群落; (b)古菌群落Figure 4. RDA of microbial community structure and environmental factors (red circles: dominant families; black arrows: the environmental factors; arrow length: the magnitude of the environmental factors; arrow direction: the correlation between the environmental factors and the microbes): (a) bacterial community; (b) archaeal community在古菌群落中,氢营养型产甲烷菌甲烷杆菌科(Methanobacteriaceae)主要分布在高COD’的环境中[35],但与苯酚相关性较弱,说明葡萄糖降解过程的氢气产量更多. Woesearchaeale与苯酚呈明显正相关,但是与COD’相关性较弱,宏基因组分析推测该菌群具有乙酸代谢能力[36]. Methanofastidiosaceae,Micrarchaeales和Bathyarchaeia的分布特征较为相似[37−39],均趋向于分布与COD’较低的环境中,苯酚对其影响较小,目前推测3个菌科具有产甲烷代谢过程. Lokiarchaeia、Asgardarchaeota及未分类菌科(Unassigned)的分布与苯酚和COD’均呈负相关关系,说明有其他环境因子影响该菌科的分布.
2.5 厌氧降解产物分析
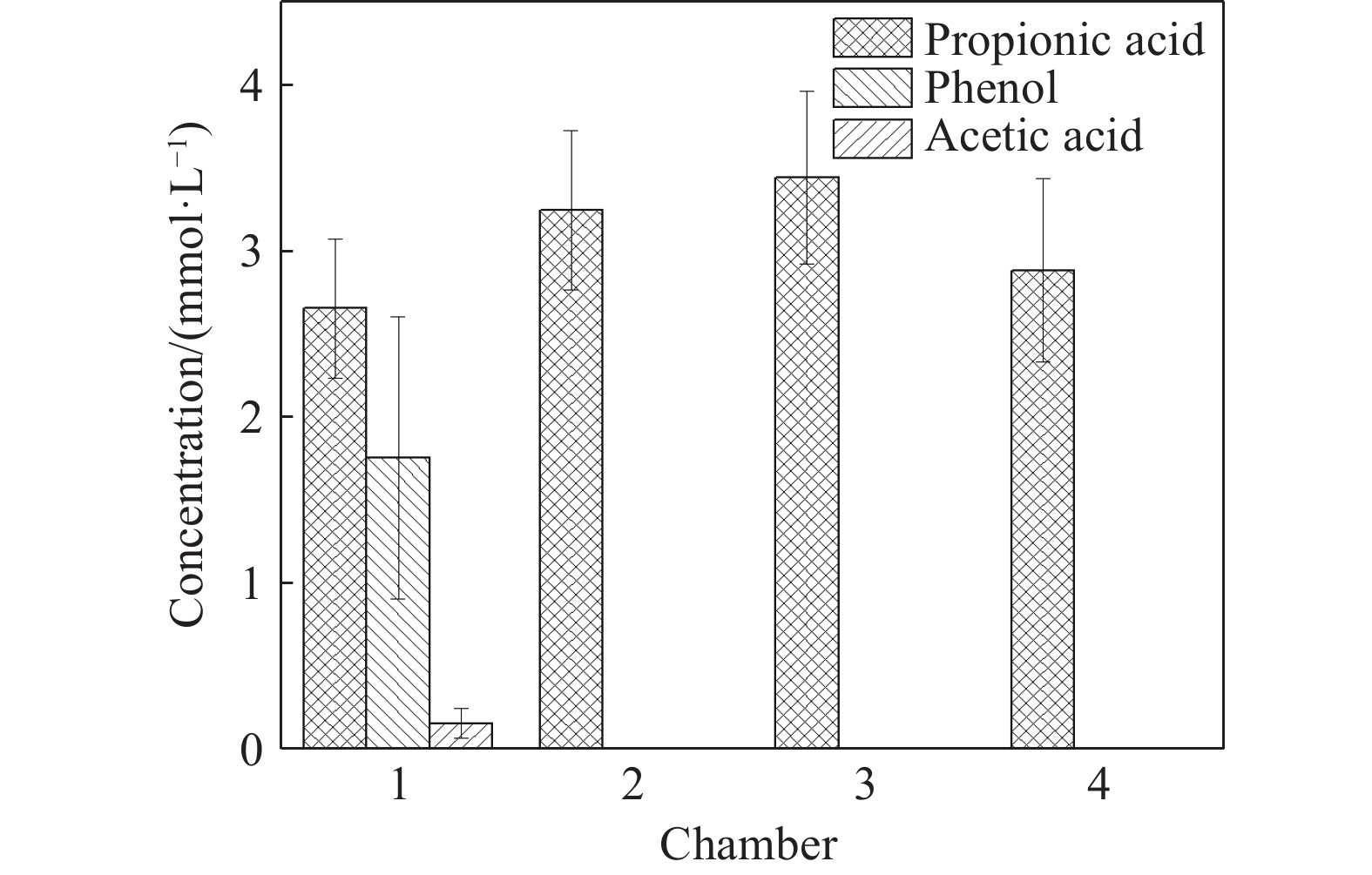
定性分析共检测出4种代谢产物,其基本信息如表3所示. 其中乙酸、苯甲酸及环己烷羧酸仅在第1隔室中检出,而丙酸在所以隔室中均被检出. 已有研究结果表明,乙酸、苯甲酸和环己烷羧酸是细菌群落优势种互营菌科(Syntrophaceae)的代谢产物,但目前仅有少量研究确认丙酸在苯酚厌氧降解过程中产生,与之相关的代谢通路尚未见报导[40−42].
表 3 苯酚厌氧降解产物Table 3. Overview of the identified intermediates in phenol’s anaerobic biodegradationCompound Chemical abstracts service
registry number (CAS#)Retention
time/minAcetic acid 64-19-7 3.177 Propionic acid 79-09-4 3.975 Cyclohexane carboxylic acid 98-89-5 10.407 Benzoic acid 65-85-0 14.230 代谢产物定量分析结果如图5所示. 在第1隔室中乙酸和苯酚浓度保持在较低的水平,至第2隔室苯酚已完全降解. 然而丙酸浓度在所有隔室中始终保持在较高水平,仅在第4隔室中有所下降. 由此推断丙酸是导致第IV阶段出水pH和COD波动的原因. 在常温厌氧条件下,降解反应的热力学条件会对丙酸的积累有重要的影响,因此需要对丙酸降解反应的吉布斯自由能变化情况进行分析.
2.6 丙酸降解反应的吉布斯自由能变化
在常温厌氧条件下,丙酸可以通过smithella途径或methylmalonyl CoA途径降解[43],其反应方程如表4所示. 气体分析结果显示第1隔室中氢气分压为20 Pa,此时反应1和2的吉布斯自由能变化量(ΔG’)值均大于−20 kJ·mol−1,说明丙酸降解反应并不能进行[44]. 在该隔室内具有丙酸降解能力的互营杆菌 (Syntrophobacteracea)和Smithellaceae菌生长受到抑制,其丰度仅为0.35%. 在后续隔室中氢气分压逐渐降低至5 Pa,降解反应的热力学条件有所改善,有利于丙酸降解菌的生长. 其在第3、4隔室中的丰度也升高至5.34%和6.9%. 然而丙酸降解产生的能量较少导致相关菌种生长缓慢,最终造成最终出水的COD去除率难以进一步提升. 因此在采用ABR处理苯酚废水时可以考虑在后续隔室添加固定填料及增加菌种接种量的方式来达到更佳的处理效果.
表 4 产甲烷条件下丙酸降解反应的吉布斯自由能变化Table 4. Gibbs free energy changes in propionate oxidation during methanogenic fermentationEquation number Reaction ΔG°’/(kJ·mol−1) ΔG’/(kJ·mol−1) 1 CH3CH2COO− + 3H2O → CH3COO− + ${\mathrm{HCO}}_3^- $ + H+ + 3H2 77.4 −16.3 2 CH3CH2COO− + H2O → 1.5CH3COO− + 0.5H+ + H2 24.3 −15.7 Note: The partial pressure of H2 was 20 Pa. The concentrations of acetate, propionate, and ${\mathrm{HCO}}_3^- $ were 1.6 × 10−4, 2.66 × 10−3 and 1.4 × 10−2 mol·L−1, respectively. The reaction temperature was 303 K. The ΔG°’ is the standard Gibbs free energy change, the ΔG’ values were calculated according to Thauer et al. [44]. 3. 结论
(1) 以葡萄糖作为进水碳源时产酸反应进行剧烈,从而造成前端隔室内pH波动较大;以苯酚作为进水碳源时产酸反应进行相对缓和. 第1、2隔室降解了90%以上的COD和苯酚,第3、4隔室出水COD长期保持100 左右,说明出水含有未降解物质.
(2) 同时使用葡萄糖和苯酚作为进水碳源可以显著的提升细菌和古菌群落的α生物多样性,有利于ABR系统稳定性的提升.
(3) 苯酚驯化过程导致细菌群落的优势菌群从链球菌科和肠杆菌科逐渐转变为互营菌科;古菌群落的优势菌群从Methanobacteriaceae逐渐转变为Methanofastidiosaceae和Micrarchaeales.
(4) 细菌群落中具有葡萄糖降解能力的Streptococcaceae、Enterobacteriaceae和Atopobiaceae趋向于分布在高COD’环境,具有苯酚降解能力的互营菌科和Desulfotomaculaceae趋向于分布在高苯酚环境,这一结果与菌种在科水平的代谢特征一致. 古菌群落中的氢营养型菌科Methanobacteriaceae分布在高COD’的环境,说明葡萄糖降解过程产生的氢气更多,Woesearchaeale分布在高苯酚环境,推测与乙酸降解过程有关.
(5) 丙酸降解缓慢是导致第IV阶段出水COD去除率难以提升的原因. 热力学计算表明隔室内氢气分压的变动是影响丙酸降解的重要因素.
-
图 4 微生物群落结构与环境因子间的RDA分析(红点表示群落中的优势菌科,黑色箭头表示环境因子,箭头的长度表示环境因子的影响力,箭头方向与红点的夹角表示微生物群落结构与该环境因子的相关性). (a)细菌群落; (b)古菌群落
Figure 4. RDA of microbial community structure and environmental factors (red circles: dominant families; black arrows: the environmental factors; arrow length: the magnitude of the environmental factors; arrow direction: the correlation between the environmental factors and the microbes): (a) bacterial community; (b) archaeal community
表 1 不同阶段ABR运行条件
Table 1 Operating conditions of ABR across different phases
Stage Glucose
concentration/(g·L−1)COD equivalent of
glucose/(g·L−1)Phenol
concentration/(g·L−1)COD equivalent of
phenol/(g·L−1)Time/d I 2.00 2.02 0 0 62 II 1.75 1.75 0.10 0.26 26 III 1.24 1.24 0.30 0.77 46 IV 0 0 0.78 1.99 22 表 2 不同污泥样品的α多样性分析
Table 2 α-diversity analysis of different sludge samples
Samples Bacterial community Archaeal community Shannon Simpson Chao1 Shannon Simpson Chao1 CG.1 2.01 0.65 284 3.42 0.90 404 CG.2 3.37 0.89 230 3.96 0.92 415 CG.3 5.37 0.99 673 4.48 0.97 394 CG.4 5.13 0.98 716 4.25 0.95 632 Phe100.1 2.87 0.80 305 3.59 0.90 510 Phe100.2 3.36 0.89 317 4.29 0.95 564 Phe100.3 4.09 0.95 416 4.65 0.97 589 Phe100.4 3.92 0.92 383 4.82 0.98 486 Phe300.1 5.44 0.98 1027 3.68 0.87 1100 Phe300.2 5.70 0.97 1111 4.43 0.88 1620 Phe300.3 5.68 0.98 860 4.91 0.94 1489 Phe300.4 4.14 0.94 292 5.78 0.99 1585 Phe780.1 3.57 0.90 402 2.96 0.84 370 Phe780.2 4.46 0.95 608 4.37 0.97 458 Phe780.3 5.22 0.98 865 4.17 0.96 585 Phe780.4 5.28 0.99 807 4.45 0.97 580 表 3 苯酚厌氧降解产物
Table 3 Overview of the identified intermediates in phenol’s anaerobic biodegradation
Compound Chemical abstracts service
registry number (CAS#)Retention
time/minAcetic acid 64-19-7 3.177 Propionic acid 79-09-4 3.975 Cyclohexane carboxylic acid 98-89-5 10.407 Benzoic acid 65-85-0 14.230 表 4 产甲烷条件下丙酸降解反应的吉布斯自由能变化
Table 4 Gibbs free energy changes in propionate oxidation during methanogenic fermentation
Equation number Reaction ΔG°’/(kJ·mol−1) ΔG’/(kJ·mol−1) 1 CH3CH2COO− + 3H2O → CH3COO− + ${\mathrm{HCO}}_3^- $ + H+ + 3H2 77.4 −16.3 2 CH3CH2COO− + H2O → 1.5CH3COO− + 0.5H+ + H2 24.3 −15.7 Note: The partial pressure of H2 was 20 Pa. The concentrations of acetate, propionate, and ${\mathrm{HCO}}_3^- $ were 1.6 × 10−4, 2.66 × 10−3 and 1.4 × 10−2 mol·L−1, respectively. The reaction temperature was 303 K. The ΔG°’ is the standard Gibbs free energy change, the ΔG’ values were calculated according to Thauer et al. [44]. -
[1] Cooper R L, Wheatstone K C. The determination of phenols in aqueous effluents. Water Res, 1973, 7(9): 1375 doi: 10.1016/0043-1354(73)90014-6
[2] Fedorak P M, Hrudey S E. Anaerobic treatment of phenolic coal conversion wastewater in semicontinuous cultures. Water Res, 1986, 20(1): 113 doi: 10.1016/0043-1354(86)90222-8
[3] Nakhla G F, Suidan M T. Anaerobic toxic wastes treatment: Dilution effects. J Hazard Mater, 1995, 42(1): 71 doi: 10.1016/0304-3894(95)00007-H
[4] 李济吾, 张珍. 真菌在含酚废水处理中的应用. 环境工程学报, 2007, 1(2):20 doi: 10.3969/j.issn.1673-9108.2007.02.005 Li J W, Zhang Z. Application of fungi in treatment of phenol-containing wastewater. Chin J Environ Eng, 2007, 1(2): 20 doi: 10.3969/j.issn.1673-9108.2007.02.005
[5] 杨朋兵, 李祥, 黄勇, 等. 苯酚对厌氧氨氧化污泥脱氮效能长短期影响. 环境科学, 2015, 36(10):3771 Yang P B, Li X, Huang Y, et al. Short or long term influence of phenol on nitrogen removal efficiency of ANAMMOX sludge. Environ Sci, 2015, 36(10): 3771
[6] 赖鹏, 赵华章, 叶正芳, 等. 生物滤池A/O工艺处理焦化废水研究. 环境科学, 2007, 28(12):2727 doi: 10.3321/j.issn:0250-3301.2007.12.011 Lai P, Zhao H Z, Ye Z F, et al. Study on treatment of coking wastewater by A/O process of biological filter. Environ Sci, 2007, 28(12): 2727 doi: 10.3321/j.issn:0250-3301.2007.12.011
[7] 马鸿志, 武文宇, 于子强, 等. 微生物合成己酸的基本原理:能量代谢及影响因素. 工程科学学报, 2023, 45(4):681 Ma H Z, Wu W Y, Yu Z Q, et al. Mechanism of caproic acid biosynthesis: Energy metabolism and influencing factors. Chin J Eng, 2023, 45(4): 681
[8] Hao L P, Michaelsen T Y, Singleton C M, et al. Novel syntrophic bacteria in full-scale anaerobic digesters revealed by genome-centric metatranscriptomics. ISME J, 2020, 14(4): 906 doi: 10.1038/s41396-019-0571-0
[9] Tomei M C, Angelucci D M, Clagnan E, et al. Anaerobic biodegradation of phenol in wastewater treatment: Achievements and limits. Appl Microbiol Biotechnol, 2021, 105(6): 2195 doi: 10.1007/s00253-021-11182-5
[10] Fuchs G, Boll M, Heider J. Microbial degradation of aromatic compounds—from one strategy to four. Nat Rev Microbiol, 2011, 9: 803 doi: 10.1038/nrmicro2652
[11] 朱铁群, 李凯慧, 张杰. 活性污泥驯化的微生物生态学原理. 微生物学通报, 2008, 35(6):939 doi: 10.3969/j.issn.0253-2654.2008.06.020 Zhu T Q, Li K H, Zhang J. Microbial ecology principle of activated sludge acclimation. Microbiology, 2008, 35(6): 939 doi: 10.3969/j.issn.0253-2654.2008.06.020
[12] 杨波, 钟启俊, 李方, 等. ABR反应器处理碱减量印染废水的启动研究. 环境科学, 2013, 34(3):968 Yang B, Zhong Q J, Li F, et al. Study on the start-up of the anaerobic baffled reactor for treating alkali-deweighting and dyeing-printing wastewater. Environ Sci, 2013, 34(3): 968
[13] 姜诗慧, 彭剑峰, 宋永会, 等. 电辅助对厌氧折流板反应器处理高浓度有机废水的影响. 环境科学研究, 2015, 28(10):1610 Jiang S H, Peng J F, Song Y H, et al. Effects of electric-assiston treatment of high-strength organic wastewater in anaerobic baffled reactor. Res Environ Sci, 2015, 28(10): 1610
[14] 李慧莉, 刘紫璇, 赵红花. 厌氧折流板反应器处理废水的研究进展. 工业水处理, 2015, 35(11):5 doi: 10.11894/1005-829x.2015.35(11).005 Li H L, Liu Z X, Zhao H H. Research progress in the anaerobic baffled reactor applied to wastewater treatment. Ind Water Treat, 2015, 35(11): 5 doi: 10.11894/1005-829x.2015.35(11).005
[15] 刘然, 彭剑峰, 宋永会, 等. 厌氧折流板反应器酸化及其对微生物种群分布的影响. 环境科学, 2010, 31(7):1554 Liu R, Peng J F, Song Y H, et al. Acidification and its effect on the population distributions of microorganisms in an anaerobic baffled reactor. Environ Sci, 2010, 31(7): 1554
[16] 许培雅, 毛丹丹, 吴建江, 等. 生活污水污泥驯化过程中细菌多样性及演替规律. 浙江工业大学学报, 2012, 40(6):606 doi: 10.3969/j.issn.1006-4303.2012.06.004 Xu P Y, Mao D D, Wu J J, et al. Bacterial diversity and succession law during activated sludge acclimation within domestic wastewater treatment system. J Zhejiang Univ Technol, 2012, 40(6): 606 doi: 10.3969/j.issn.1006-4303.2012.06.004
[17] Gao D W, Tao Y. Current molecular biologic techniques for characterizing environmental microbial community. Front Environ Sci Eng, 2012, 6(1): 82 doi: 10.1007/s11783-011-0306-6
[18] 余泽晖, 梁志伟, 李浩聪, 等. 厌氧消化污泥微生物组研究方法及应用. 微生物学通报, 2019, 46(8):2053 Yu Z H, Liang Z W, Li H C, et al. Anaerobic sludge digestion microbiome—analytical methods and applications. Microbiol China, 2019, 46(8): 2053
[19] Margulies M, Egholm M, Altman W E, et al. Genome sequencing in microfabricated high-density picolitre reactors. Nature, 2005, 437(7057): 376 doi: 10.1038/nature03959
[20] Koonin E V, Wolf Y I. Genomics of bacteria and Archaea: The emerging dynamic view of the prokaryotic world. Nucleic Acids Res, 2008, 36(21): 6688 doi: 10.1093/nar/gkn668
[21] Soh Y N A, Kunacheva C, Webster R D, et al. Composition and biotransformational changes in soluble microbial products (SMPs) along an anaerobic baffled reactor (ABR). Chemosphere, 2020, 254: 126775 doi: 10.1016/j.chemosphere.2020.126775
[22] Saminathan T, García M, Ghimire B, et al. Metagenomic and metatranscriptomic analyses of diverse watermelon cultivars reveal the role of fruit associated microbiome in carbohydrate metabolism and ripening of mature fruits. Front Plant Sci, 2018, 9: 4 doi: 10.3389/fpls.2018.00004
[23] Duarte C M, Røstad A, Michoud G, et al. Discovery of Afifi, the shallowest and southernmost brine pool reported in the Red Sea. Sci Rep, 2020, 10(1): 910 doi: 10.1038/s41598-020-57416-w
[24] Bolyen E, Rideout J R, Dillon M R, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2. Nat Biotechnol, 2019, 37(8): 852 doi: 10.1038/s41587-019-0209-9
[25] Callahan B J, McMurdie P J, Rosen M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods, 2016, 13(7): 581 doi: 10.1038/nmeth.3869
[26] Yarza P, Yilmaz P, Pruesse E, et al. Uniting the classification of cultured and uncultured bacteria and Archaea using 16S rRNA gene sequences. Nat Rev Microbiol, 2014, 12(9): 635 doi: 10.1038/nrmicro3330
[27] Sillero N, Campos J C, Arenas-Castro S, et al. A curated list of R packages for ecological niche modelling. Ecol Model, 2023, 476: 110242 doi: 10.1016/j.ecolmodel.2022.110242
[28] Gupta R S, Chen W J, Adeolu M, et al. Molecular signatures for the class Coriobacteriiaand its different clades; proposal for division of the class Coriobacteriiainto the emended order Coriobacteriales, containing the emended family Coriobacteriaceaeand and Atopobiaceae fam. nov., and Eggerthellales ord. nov., containing the family Eggerthellaceae fam. nov. Int J Syst Evol Microbiol, 2013, 63(9): 3379
[29] Gray N D, Sherry A, Grant R J, et al. The quantitative significance of Syntrophaceae and syntrophic partnerships in methanogenic degradation of crude oil alkanes. Environ Microbiol, 2011, 13(11): 2957 doi: 10.1111/j.1462-2920.2011.02570.x
[30] Hanselmann K W, Kaiser J P, Wenk M, et al. Growth on methanol and conversion of methoxylated aromatic substrates by Desulfotomaculum orientis in the presence and absence of sulfate. Microbiol Res, 1995, 150(4): 387 doi: 10.1016/S0944-5013(11)80021-2
[31] Cupples A M. The use of nucleic acid based stable isotope probing to identify the microorganisms responsible for anaerobic benzene and toluene biodegradation. J Microbiol Meth, 2011, 85(2): 83 doi: 10.1016/j.mimet.2011.02.011
[32] Liang B, Wang L Y, Mbadinga S M, et al. Anaerolineaceae and Methanosaeta turned to be the dominant microorganisms in alkanes-dependent methanogenic culture after long-term of incubation. AMB Express, 2015, 5(1): 117
[33] Guieysse B, Wuertz S. Metabolically versatile large-genome prokaryotes. Curr Opin Biotechnol, 2012, 23(3): 467 doi: 10.1016/j.copbio.2011.12.022
[34] Waite D W, Chuvochina M, Pelikan C, et al. Proposal to reclassify the proteobacterial classes Deltaproteobacteria and Oligoflexia, and the phylum Thermodesulfobacteria into four phyla reflecting major functional capabilities. Int J Syst Evol Microbiol, 2020, 70(11): 5972 doi: 10.1099/ijsem.0.004213
[35] Granada C E, Hasan C, Marder M, et al. Biogas from slaughterhouse wastewater anaerobic digestion is driven by the archaeal family Methanobacteriaceae and bacterial families Porphyromonadaceae and Tissierellaceae. Renew Energy, 2018, 118: 840
[36] Liu X B, Li M, Castelle C J, et al. Insights into the ecology, evolution, and metabolism of the widespread Woesearchaeotal lineages. Microbiome, 2018, 6(1): 102 doi: 10.1186/s40168-018-0488-2
[37] Nobu M K, Narihiro T, Kuroda K, et al. Chasing the elusive Euryarchaeota class WSA2: Genomes reveal a uniquely fastidious methyl-reducing methanogen. ISME J, 2016, 10(10): 2478 doi: 10.1038/ismej.2016.33
[38] Chen L X, Méndez-García C, Dombrowski N, et al. Metabolic versatility of small archaea Micrarchaeota and Parvarchaeota. ISME J, 2018, 12(3): 756
[39] Romano R G, Bendia A G, Moreira J C F, et al. Bathyarchaeia occurrence in rich methane sediments from a Brazilian ría. Estuar Coast Shelf Sci, 2021, 263: 107631 doi: 10.1016/j.ecss.2021.107631
[40] Keith C L, Bridges R L, Fina L R, et al. The anaerobic decomposition of benzoic acid during methane fermentation. Arch Microbiol, 1978, 118(2): 173 doi: 10.1007/BF00415726
[41] Pullammanappallil P C, Chynoweth D P, Lyberatos G, et al. Stable performance of anaerobic digestion in the presence of a high concentration of propionic acid. Bioresour Technol, 2001, 78(2): 165 doi: 10.1016/S0960-8524(00)00187-5
[42] Poirier S, Bize A, Bureau C, et al. Community shifts within anaerobic digestion microbiota facing phenol inhibition: Towards early warning microbial indicators? Water Res, 2016, 100: 296
[43] Leng L, Yang P X, Singh S, et al. A review on the bioenergetics of anaerobic microbial metabolism close to the thermodynamic limits and its implications for digestion applications. Bioresour Technol, 2018, 247: 1095 doi: 10.1016/j.biortech.2017.09.103
[44] Thauer R K, Jungermann K, Decker K. Energy conservation in chemotrophic anaerobic bacteria. Bacteriol Rev, 1977, 41(1): 100 doi: 10.1128/br.41.1.100-180.1977



 下载:
下载: